data_count |>
reactable::reactable()Déterminer la taille des échantillons par site
On compte le nombre de mesures pour chaque combinaison.
source("src/itgr_measurements.R")
contaminants <- itgr_measurements()
data_count <- contaminants |>
dplyr::mutate(Year = as.integer(Year)) |>
toxbox::uncensored(cols = "value", keep_cens = TRUE) |>
dplyr::group_by(Location, Year, Species, conpound_family, variable) |>
dplyr::summarise(n = dplyr::n(), n_censored = sum(value_censored))Warning: There was 1 warning in `dplyr::mutate()`.
ℹ In argument: `dplyr::across(tidyselect::all_of(cols), remove_cens)`.
Caused by warning:
! NAs introduced by coercion`summarise()` has grouped output by 'Location', 'Year', 'Species',
'conpound_family'. You can override using the `.groups` argument.Criteria 1. On retire les sites pour lesquelles, il y a une seule année de collecte (impossibilité de calibrer un modèle de tendance temporelle).
Voici la liste des sites pour lesquelles, on a une seule année de mesure
crit1 <- contaminants |>
dplyr::select(Location, Year) |>
dplyr::distinct() |>
dplyr::group_by(Location) |>
dplyr::count() |>
dplyr::filter(n == 1) |>
dplyr::pull(Location)
cat(paste("*", crit1), sep="\n")- Archipel de Mingan (approximative location)
- Beauharnois
- Betchouanes
- Bonaventure
- Deslauriers
- Escuminac
- Gros Pot
- Ile Beauséjour
- Ile Bellechasse
- Ile Bienville
- Ile Blanche
- Ile Dodens
- Ile Dowker
- Ile Nue
- Ile Saint-Barnabé
- Ile a Bouleau de Terre
- Ile aux Pommes
- Ile de la Madeleine
- Ile de la Mine
- Ile du Petit Caoui
- Ile du Petit Caouis
- Ile à Calculot des Betchouanes
- Iles aux Loups Marins
- Iles de la Madeleine
- Ilets Boisés
- Ilets De Quen
- Kamouraska
- Kukuministuk (Caye à Cochons)
- Lac Bleu
- Lac Evans
- Lac Saint-Bernard
- Lac St-Pierre
- Lac à la Carpe
- Long Island
- Miller island
- Missipinuk (Grosse Romaine)
- Petit Lac Jacques Cartier
- Petite Ile Ste-Geneviève
- Pointe Saint-Pancrace
- Prairie du Castor
- Riviere George
On retire ces sites pour l’étude de la taille de l’échantillon
data_count <- data_count |>
dplyr::filter(!(Location %in% crit1)) |>
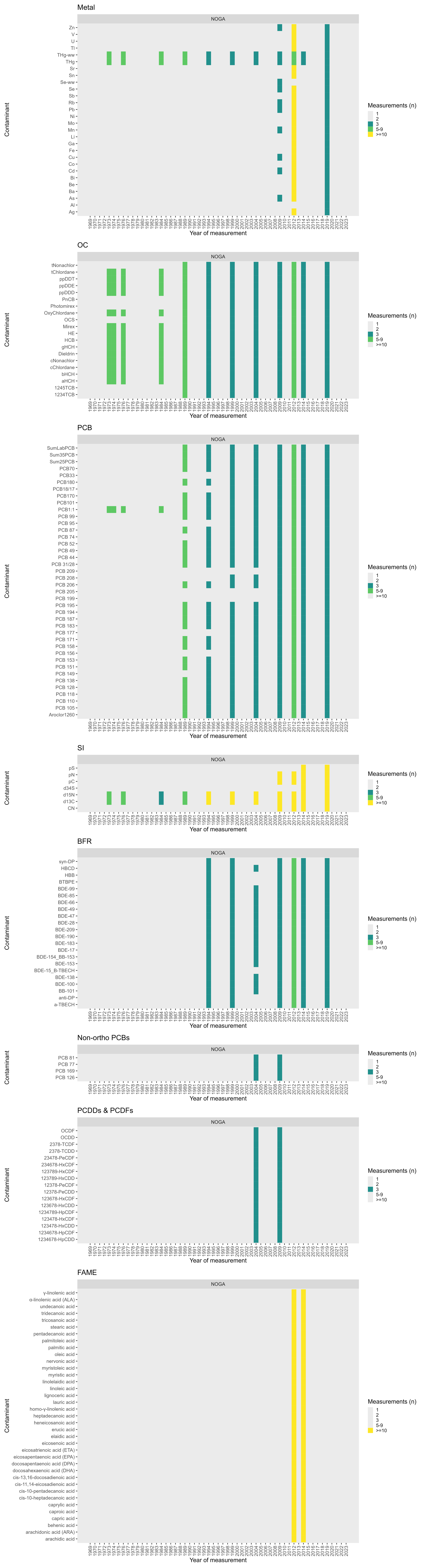
dplyr::ungroup()Site Battures aux Loups Marins
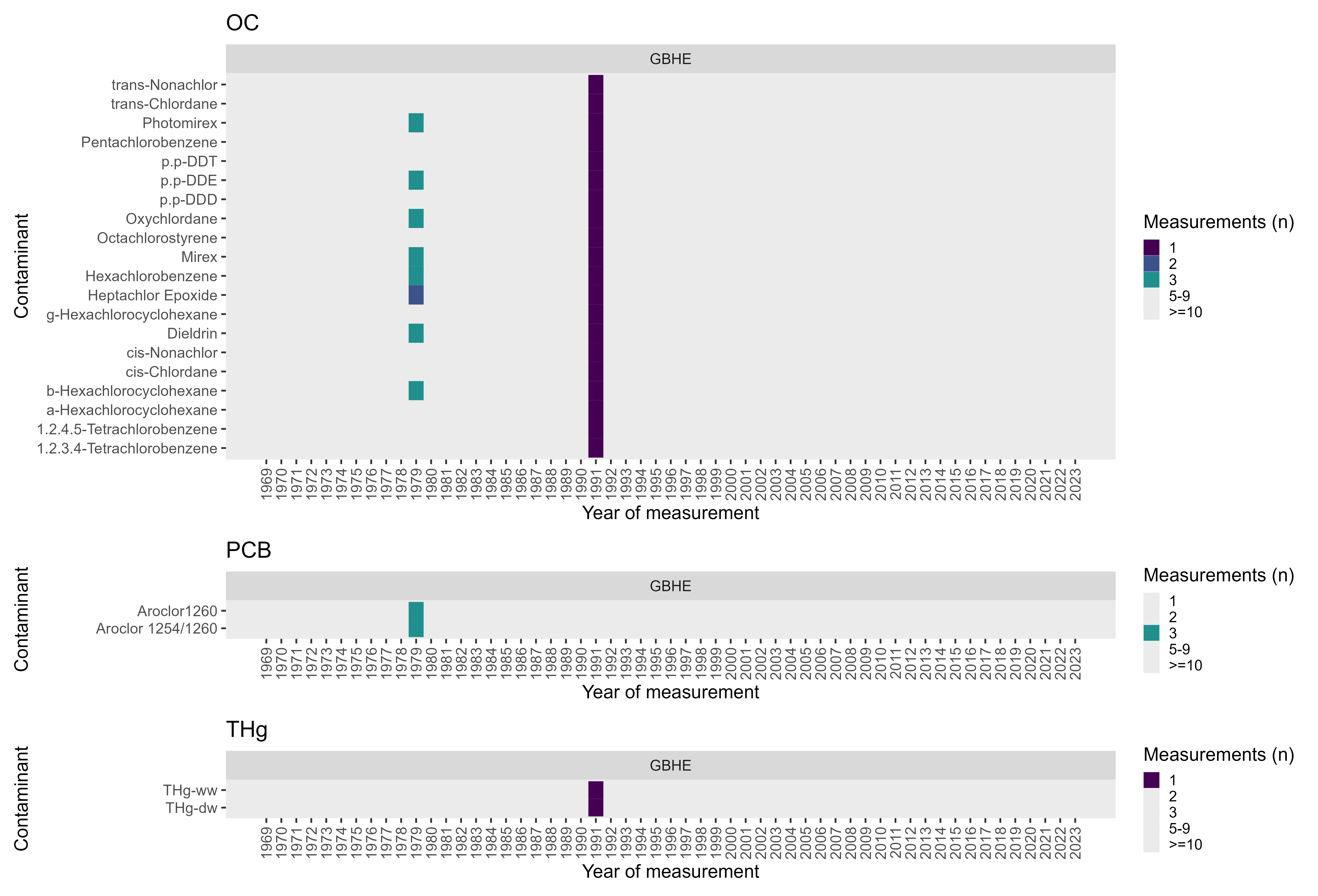
Site Grande Ile

Site Ile Carillon
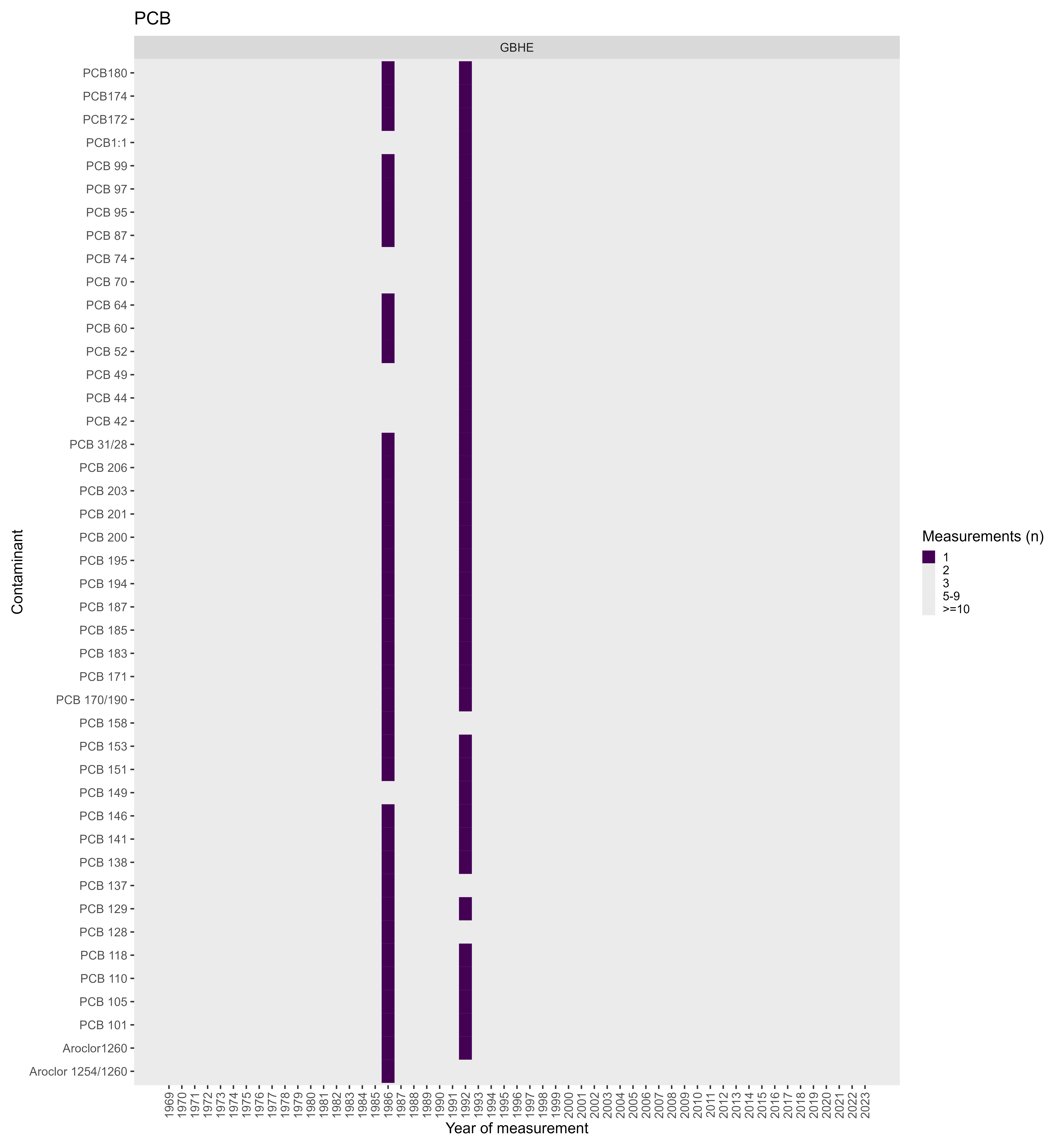
Site Ile Dickerson
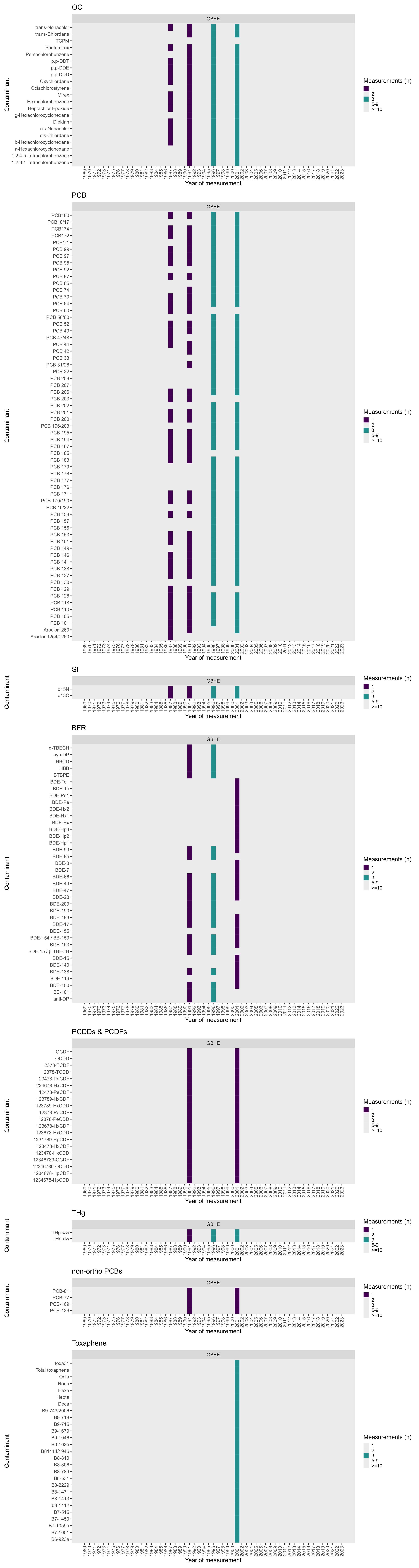
Site Ile Eaton
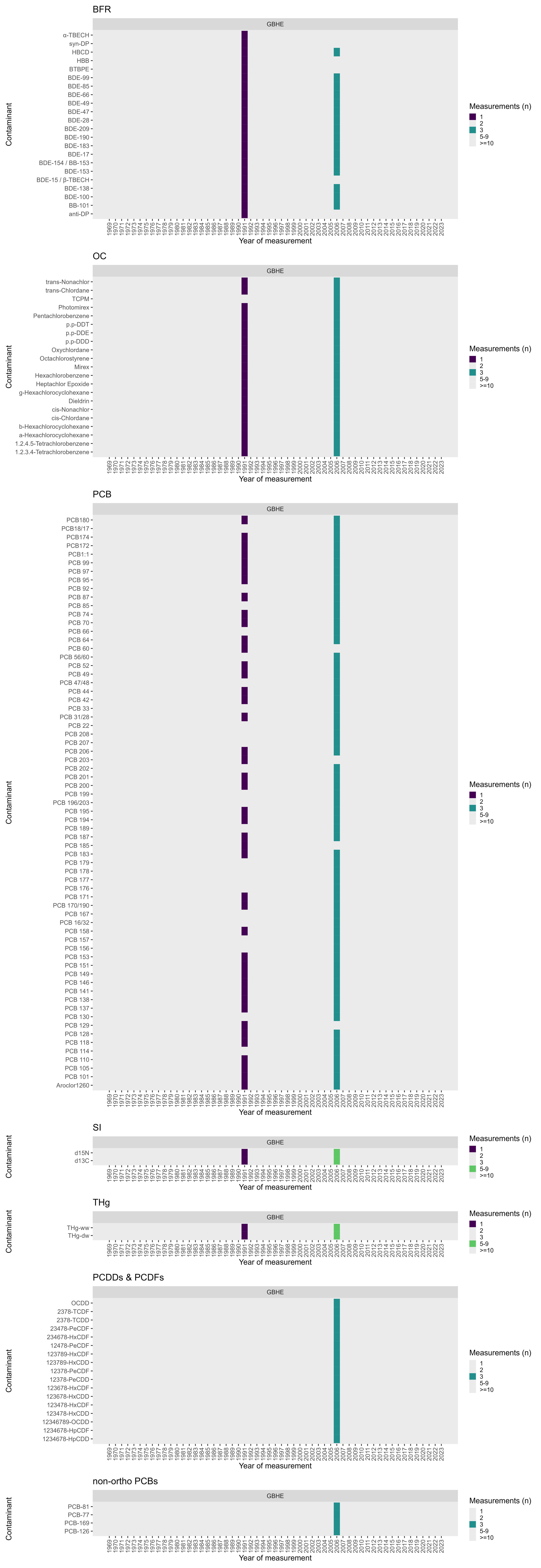
Site Ile Laval
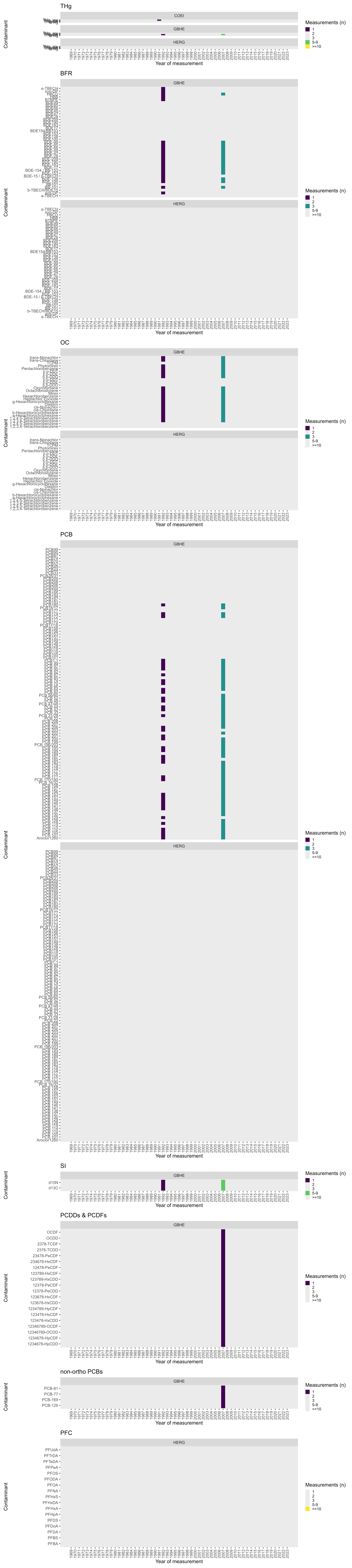
Site Ile Manowin
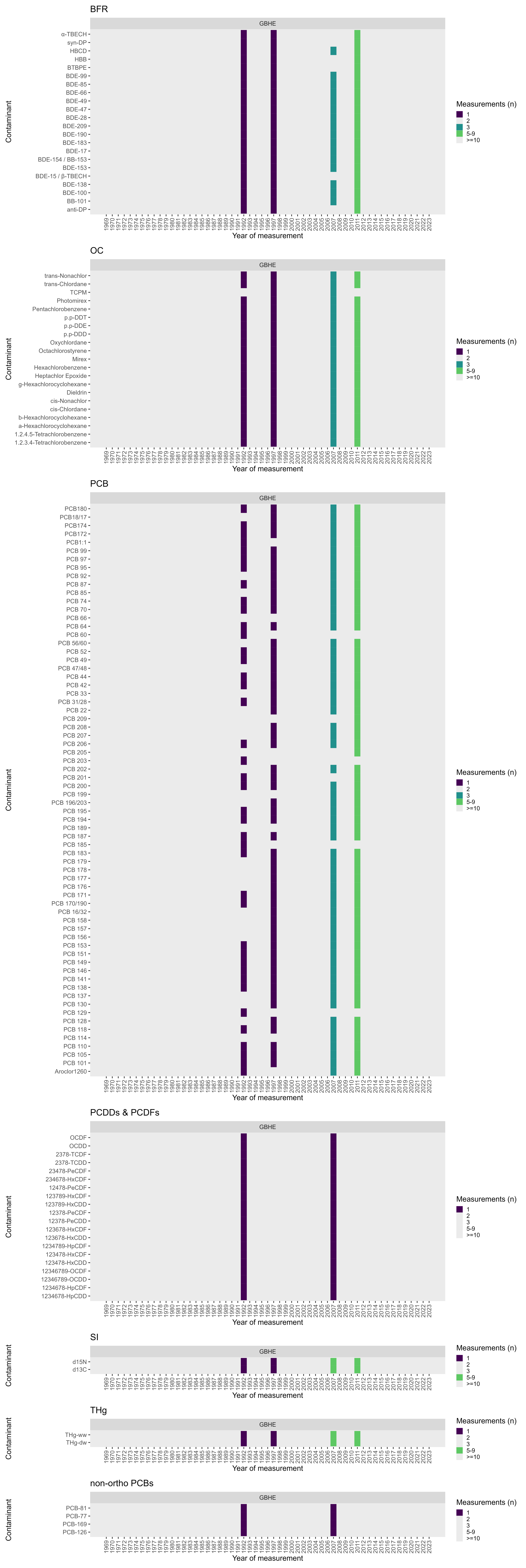
Site Ile Matane

Site Ile Saint-Bernard
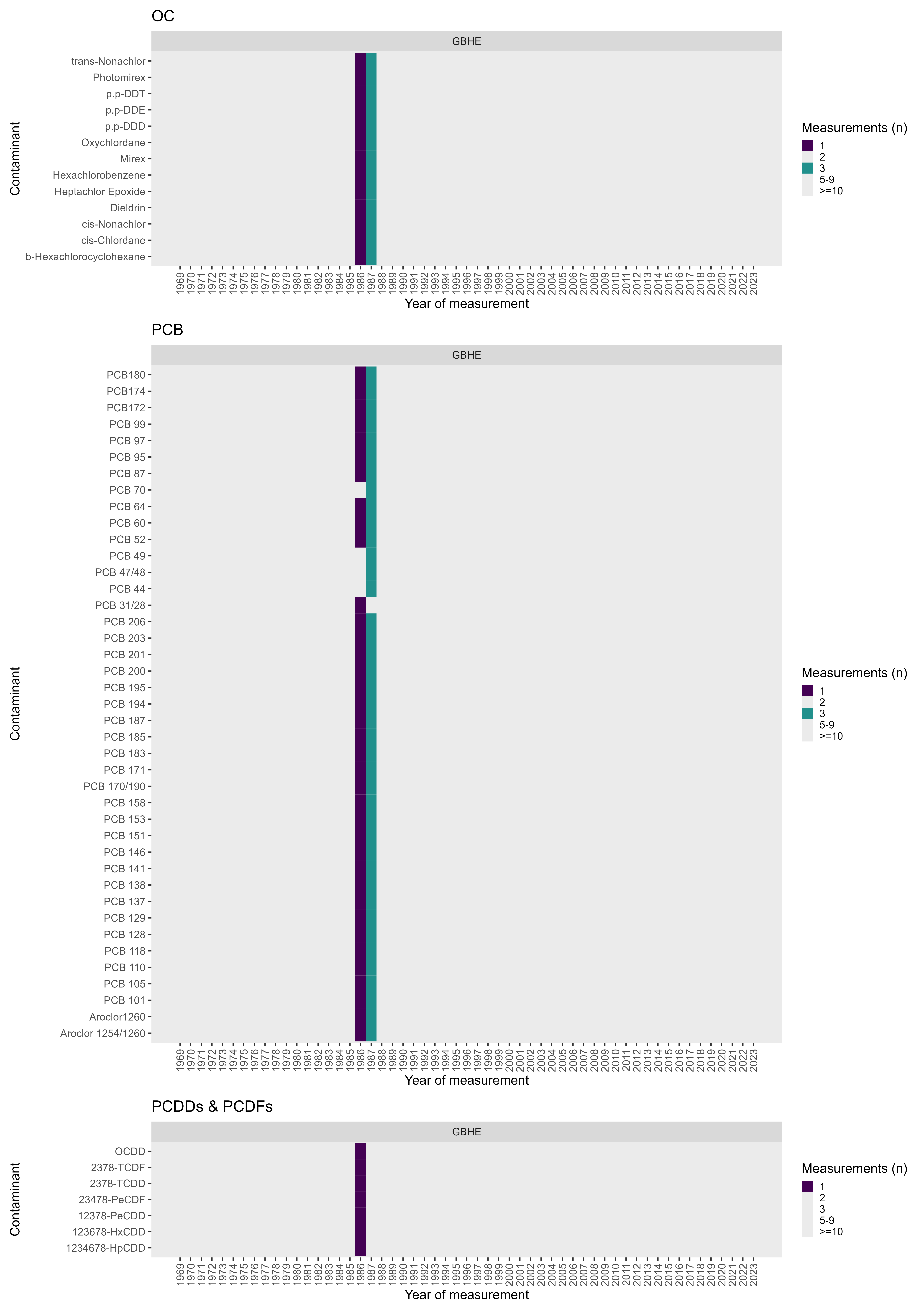
Site Ile Steamboat
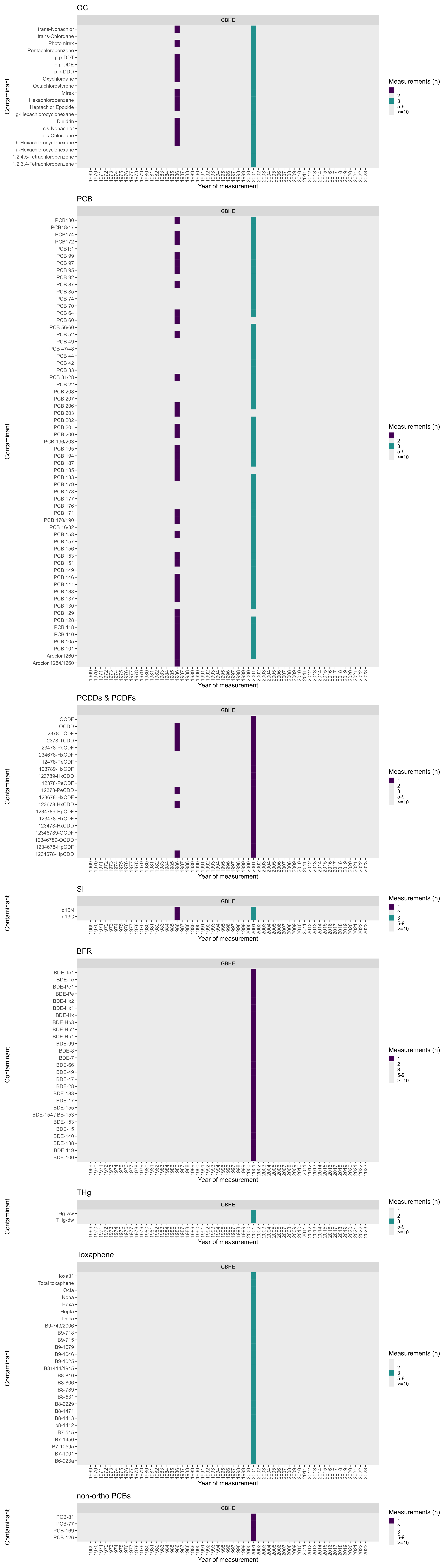
Site Ile Villemomble
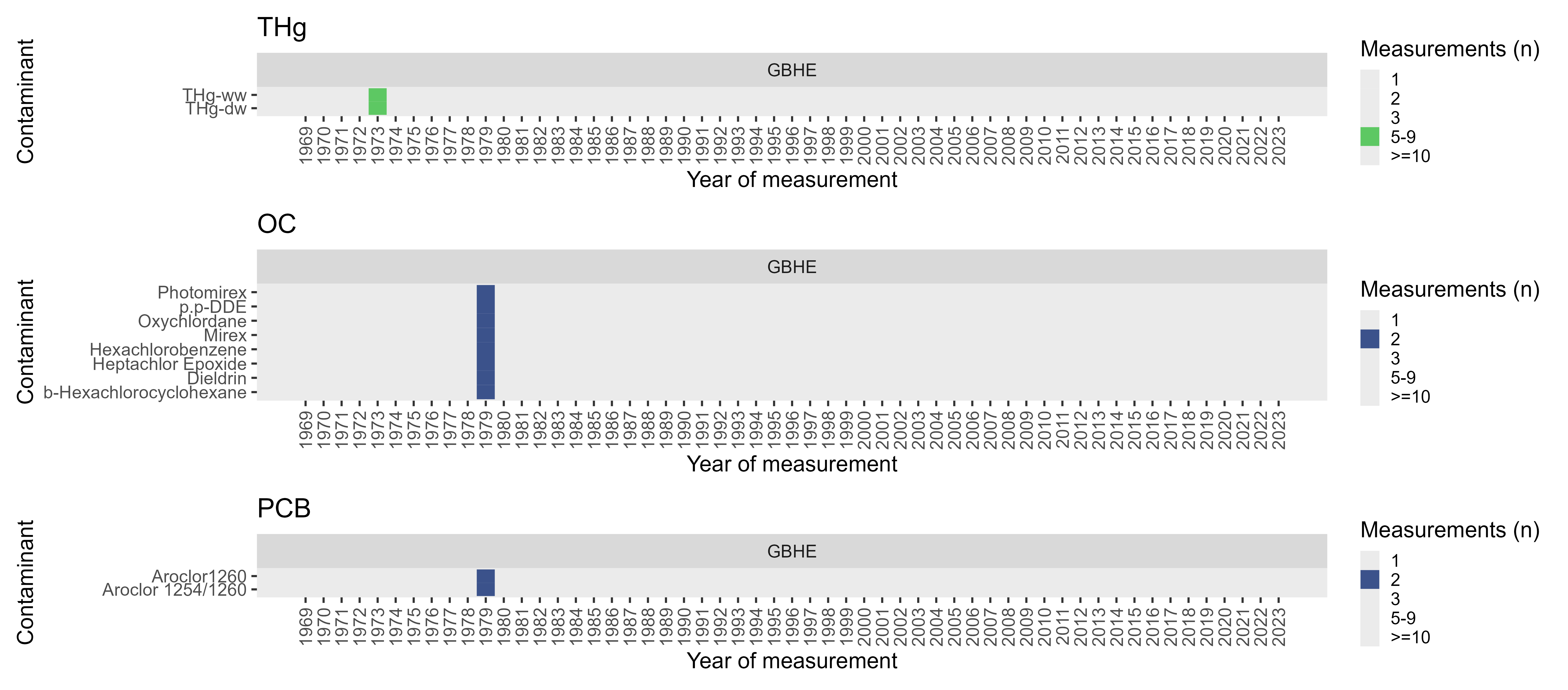
Site Ile aux Basques
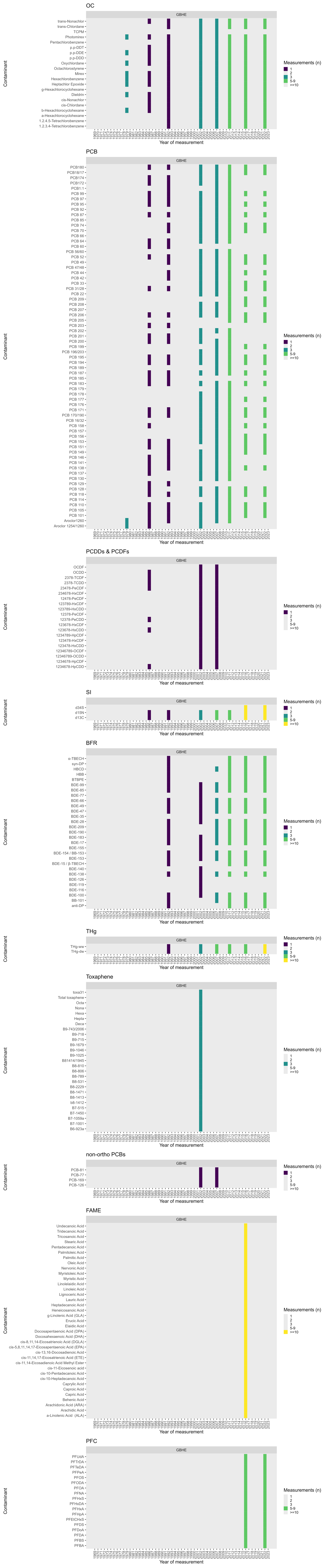
Site Ile aux Herons
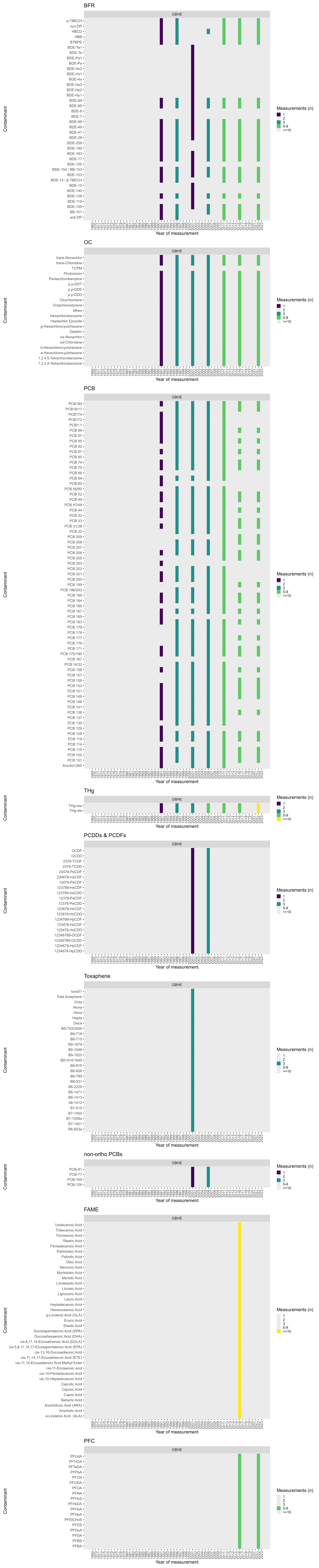
Site Ile aux Hérons

Site Ile aux Oeufs
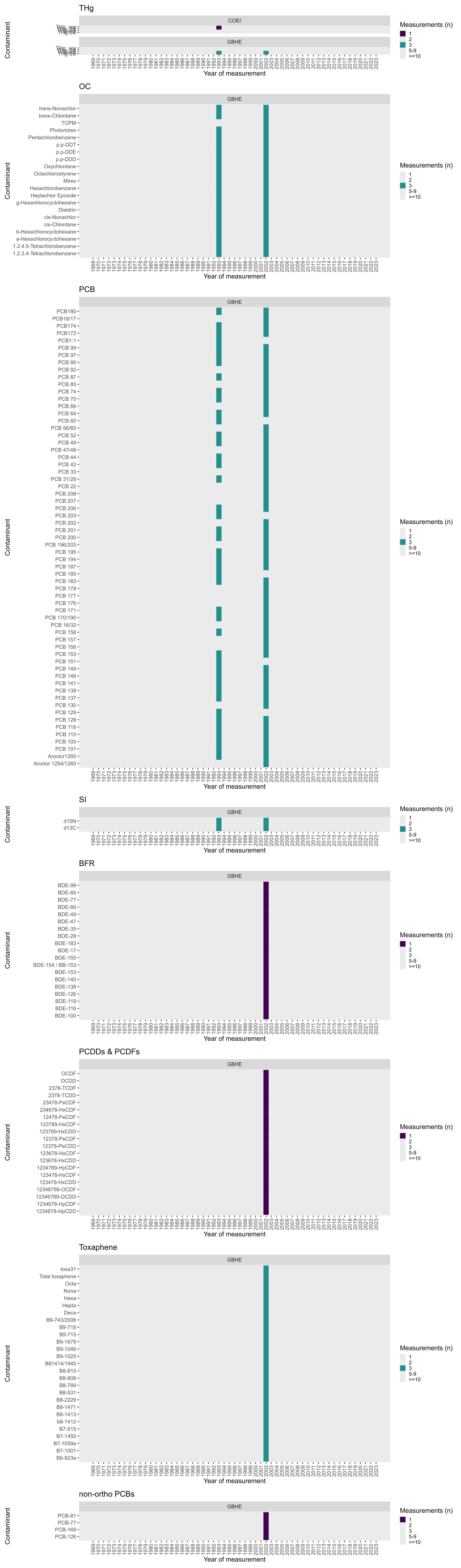
Site Ile de Carillon
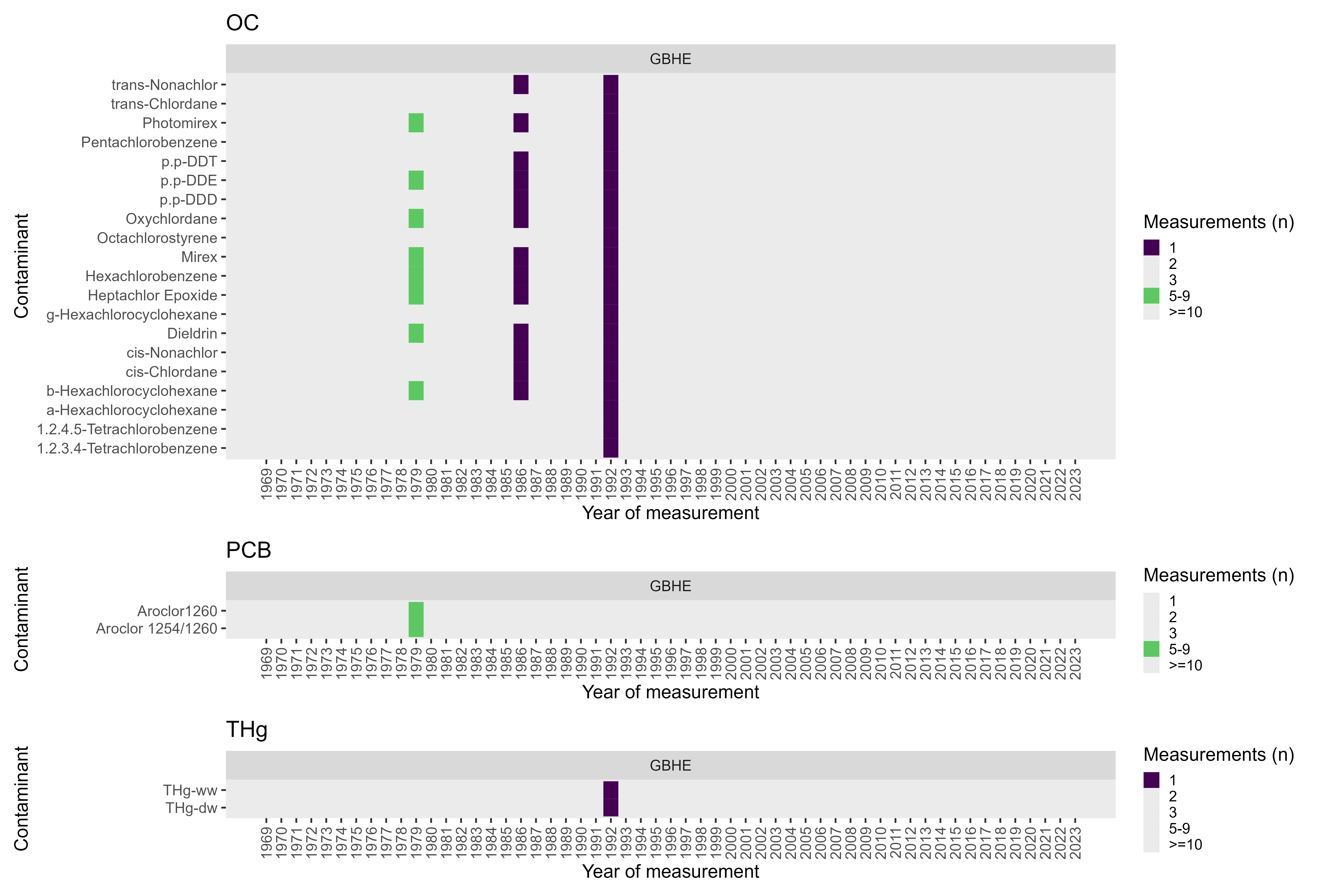
Site Ile de la Corneille

Site Ile du Bic
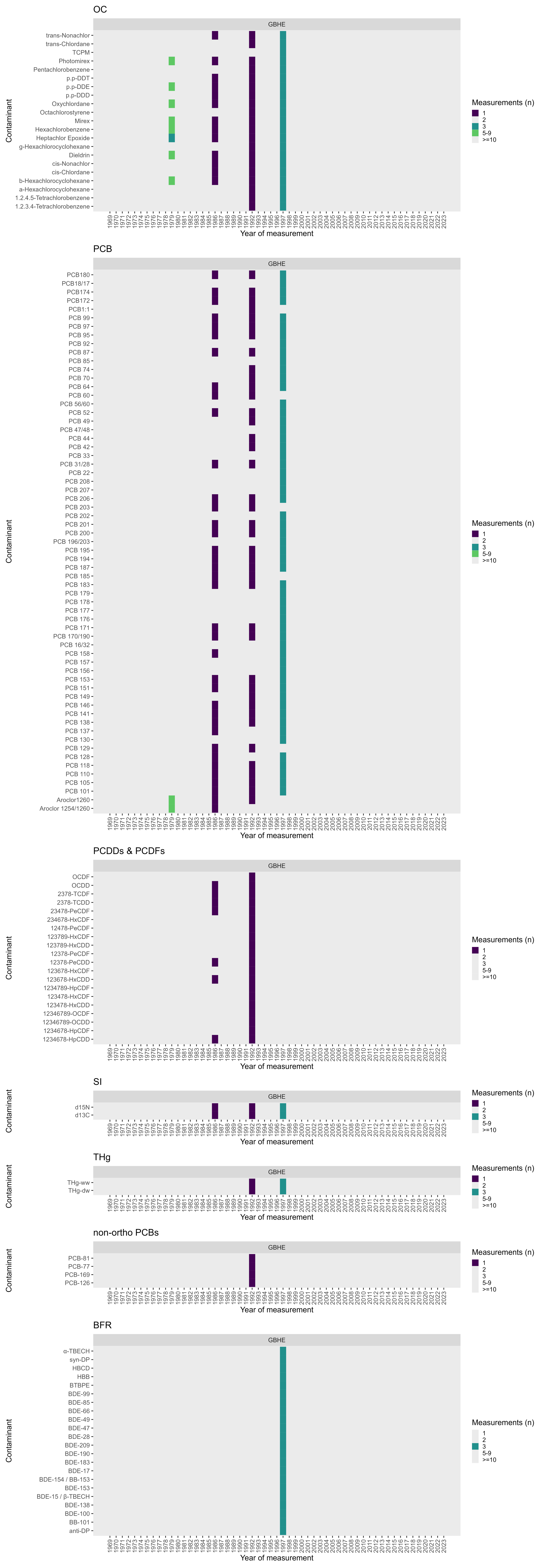
Site Ile du Corossol
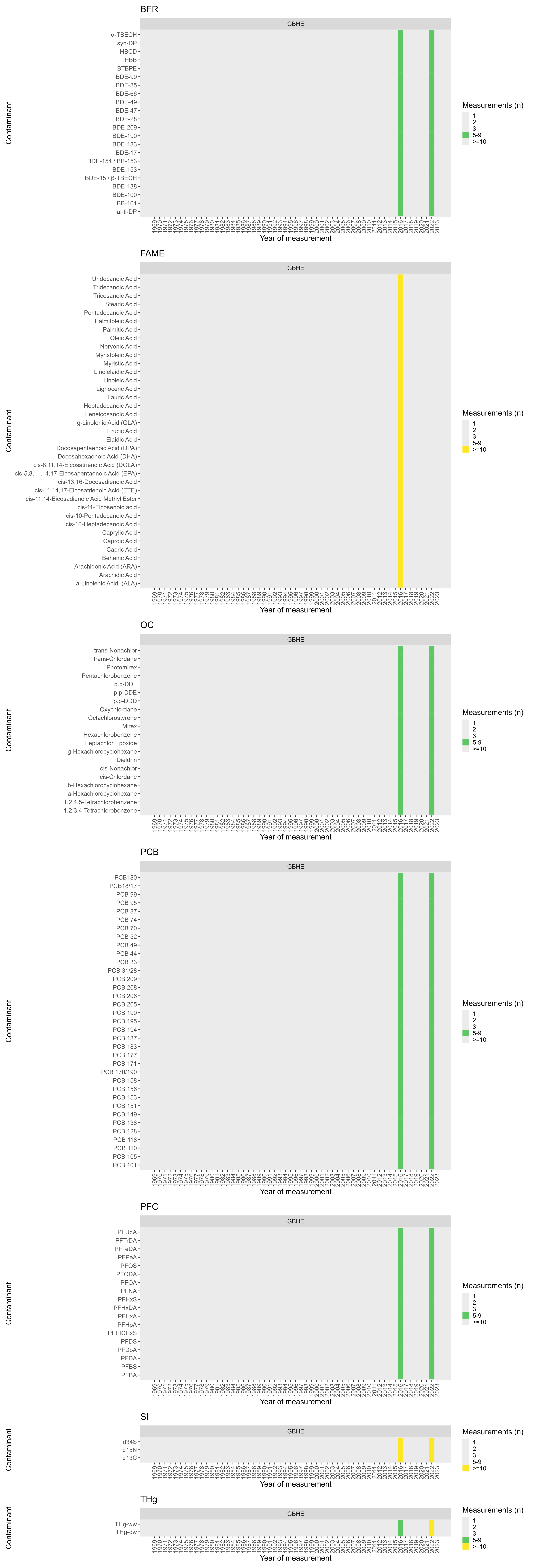
Site Iles Sainte-Marie
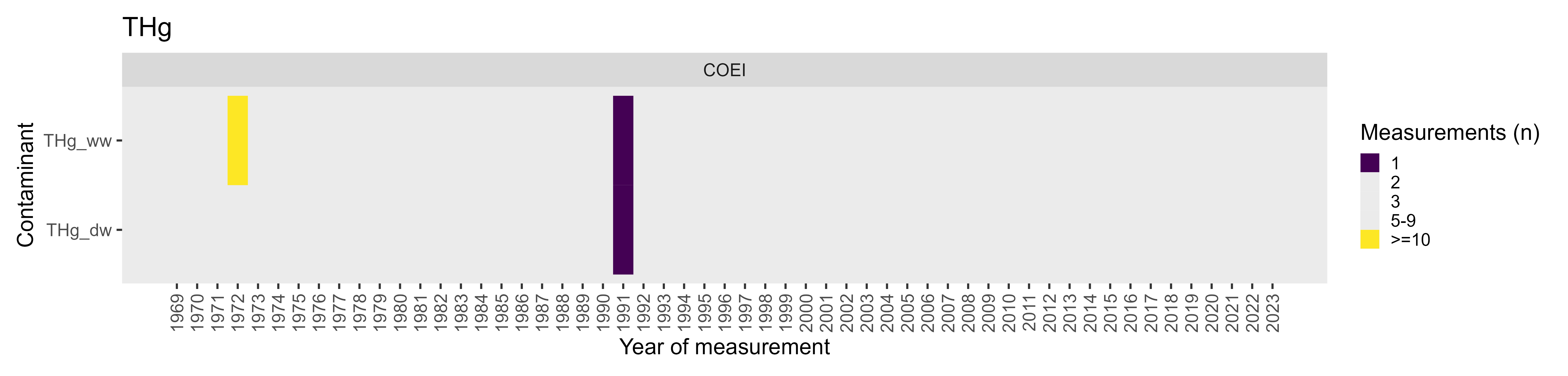
Site Maria
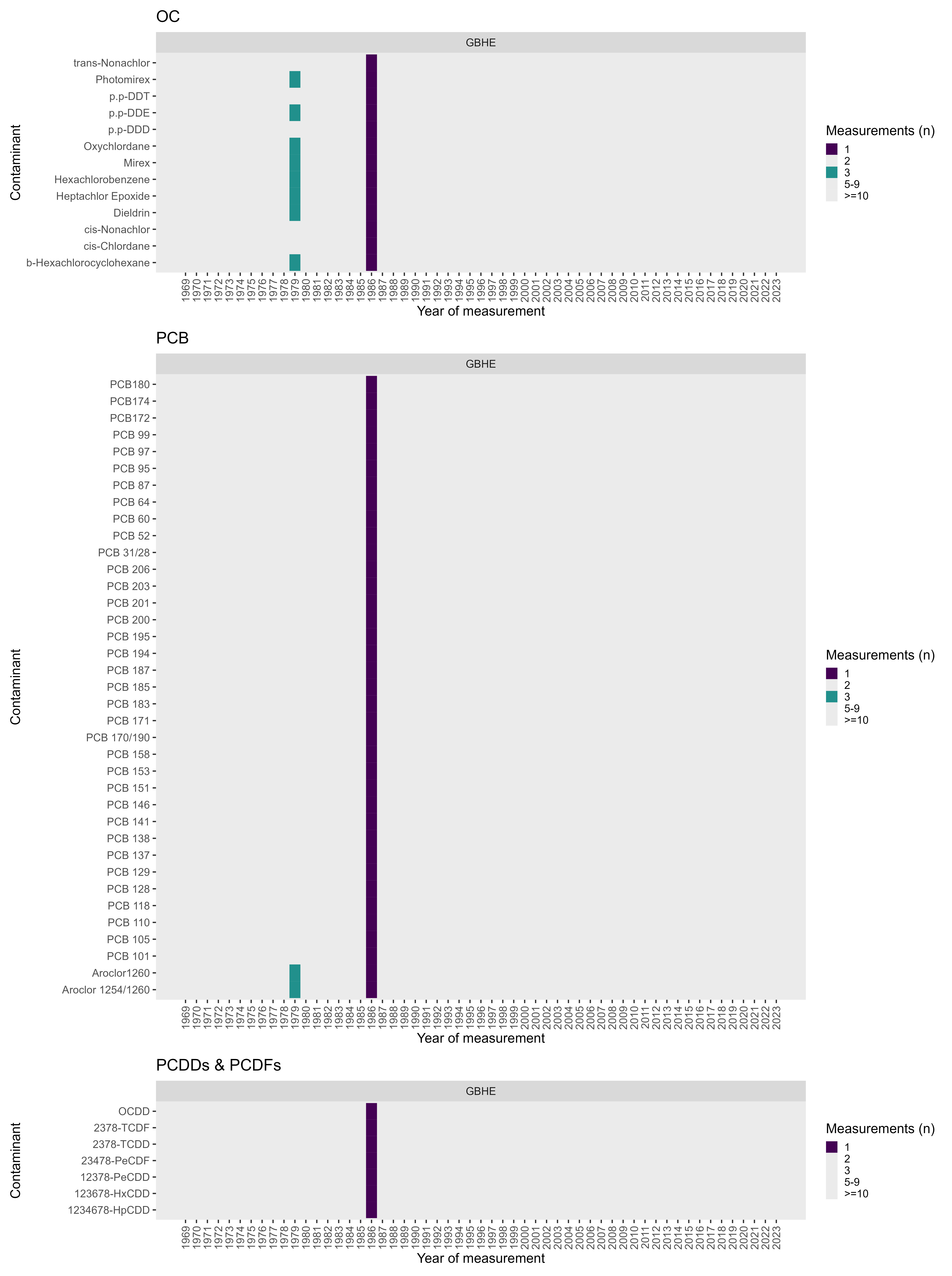
Site Petit lac Jacques-Cartier

Site Refuge Watshishou
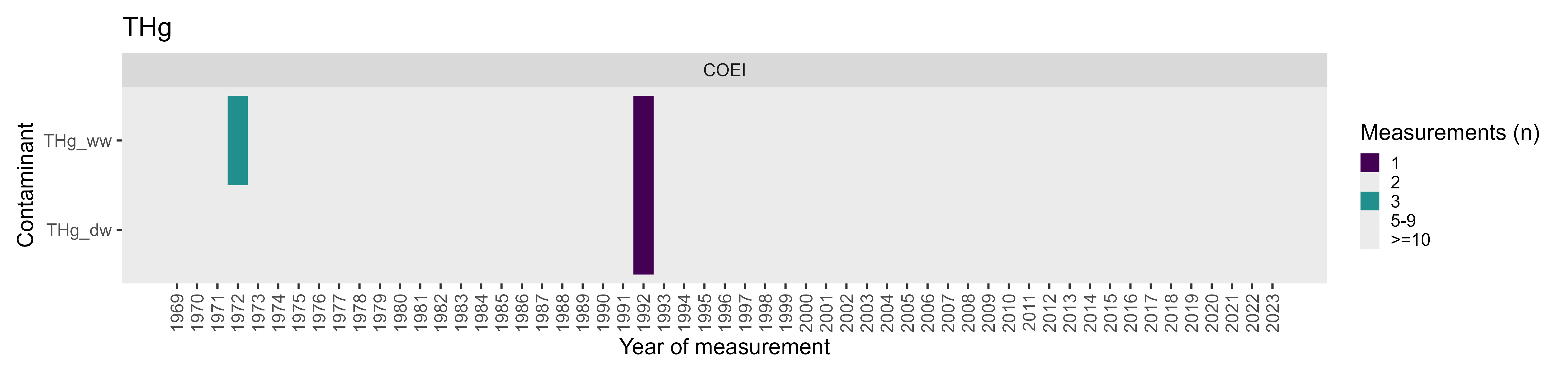
Site Yamachiche